
Funkcje, skład i budowa nukleosomów
Plik nukleosom jest to podstawowa jednostka pakowania DNA w organizmach eukariotycznych. Jest to zatem najmniejszy element kompresyjny chromatyny.
Nukleosom jest zbudowany jako oktamer białek zwanych histonami lub strukturę w kształcie bębna, na którą nawinięto około 140 nt DNA, wykonując prawie dwa pełne obroty..
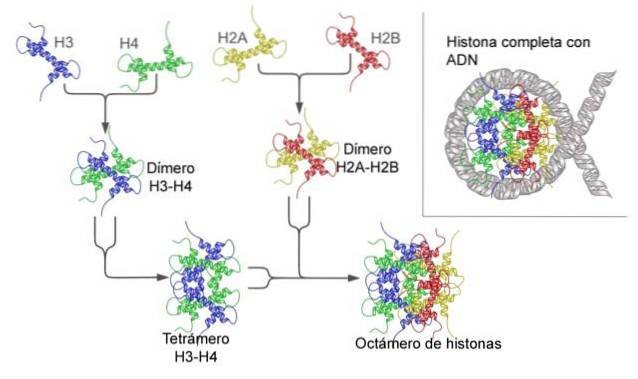
Dodatkowo uważa się, że dodatkowe 40-80 nt DNA jest częścią nukleosomu i jest to frakcja DNA, która umożliwia fizyczną ciągłość między jednym nukleosomem a drugim w bardziej złożonych strukturach chromatyny (takich jak włókno chromatynowe 30 nm)..
Kod histonowy był jednym z pierwszych molekularnie zrozumiałych elementów kontroli epigenetycznej..
Indeks artykułów
- 1 Funkcje
- 2 Skład i struktura
- 3 Zagęszczanie chromatyny
- 4 Kod histonowy i ekspresja genów
- 5 Euchromatyna vs heterochromatyna
- 6 Inne funkcje
- 7 Referencje
funkcje
Nukleosomy pozwalają:
- Pakowanie DNA, aby zmieściło się w ograniczonej przestrzeni jądra.
- Określają podział między wyrażaną chromatyną (euchromatyną) a cichą chromatyną (heterochromatyną).
- Zorganizuj całą chromatynę zarówno przestrzennie, jak i funkcjonalnie w jądrze.
- Stanowią one substrat modyfikacji kowalencyjnych, które determinują ekspresję i poziom ekspresji genów kodujących białka poprzez tak zwany kod histonowy.
Skład i struktura
W najbardziej podstawowym sensie nukleosomy składają się z DNA i białek. DNA może być praktycznie dowolnym dwupasmowym DNA obecnym w jądrze komórki eukariotycznej, podczas gdy wszystkie białka nukleosomalne należą do zestawu białek zwanych histonami..
Histony to małe białka z dużym ładunkiem reszt aminokwasowych zasadowych; Umożliwia to przeciwdziałanie wysokiemu ujemnemu ładunkowi DNA i ustanowienie wydajnego fizycznego oddziaływania między dwiema cząsteczkami bez osiągania sztywności kowalencyjnego wiązania chemicznego..
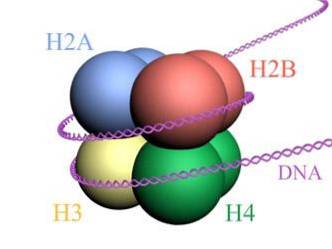
Histony tworzą podobny do bębna oktamer z dwiema kopiami lub monomerami każdego z histonów H2A, H2B, H3 i H4. DNA wykonuje prawie dwa pełne obroty po bokach oktameru, a następnie kontynuuje z ułamkiem DNA łącznika, który łączy się z histonem H1, aby powrócić i dać dwa pełne obroty na innym oktameru histonu..
Zestaw oktameru, powiązany DNA i odpowiadający mu DNA łącznikowy to nukleosom.
Zagęszczanie chromatyny
DNA genomowe składa się z niezwykle długich cząsteczek (więcej niż metr w przypadku ludzi, biorąc pod uwagę wszystkie ich chromosomy), które muszą być zagęszczone i zorganizowane w niezwykle małym jądrze.
Pierwszy krok w tym zagęszczaniu odbywa się poprzez tworzenie nukleosomów. Tylko na tym etapie DNA jest zagęszczane około 75 razy.
Daje to początek liniowemu włóknu, z którego zbudowane są kolejne poziomy zagęszczenia chromatyny: włókno 30 nm, pętle i pętle pętli..
Kiedy komórka dzieli się w wyniku mitozy lub mejozy, ostatecznym stopniem zagęszczenia jest odpowiednio sam chromosom mitotyczny lub mejotyczny..
Kod histonów i ekspresja genów
Fakt, że oktamery histonów i DNA oddziałują elektrostatycznie, częściowo wyjaśnia ich efektywną asocjację, bez utraty płynności wymaganej do wytworzenia dynamicznych elementów nukleosomów do zagęszczania i rozkładania chromatyny..
Ale jest jeszcze bardziej zaskakujący element interakcji: N-końcowe końce histonów są odsłonięte na zewnątrz wnętrza oktameru, które jest bardziej zwarte i obojętne..
Końce te nie tylko fizycznie oddziałują z DNA, ale także podlegają serii kowalencyjnych modyfikacji, od których będzie zależał stopień zagęszczenia chromatyny i ekspresja związanego z nią DNA..
Zbiór modyfikacji kowalencyjnych, między innymi pod względem rodzaju i liczby, jest zbiorczo nazywany kodem histonowym. Modyfikacje te obejmują fosforylację, metylację, acetylację, ubikwitynację i sumoilację reszt argininy i lizyny na N-końcach histonów..
Każda zmiana, wraz z innymi w tej samej cząsteczce lub w resztach innych histonów, szczególnie histonów H3, będzie determinować ekspresję lub nie związanego z nią DNA, jak również stopień zagęszczenia chromatyny..
Jako ogólną zasadę zaobserwowano na przykład, że hipermetylowane i hipoacetylowane histony decydują o tym, że powiązany DNA nie jest wyrażany i że chromatyna jest obecna w bardziej zwartym stanie (heterochromatyczny, a zatem nieaktywny)..
Natomiast euchromatyczny DNA (mniej zwarty i aktywny genetycznie) jest powiązany z chromatyną, której histony są hiperacetylowane i hipometylowane..
Euchromatyna vs heterochromatyna
Widzieliśmy już, że status modyfikacji kowalencyjnej histonów może determinować stopień ekspresji i miejscowe zagęszczenie chromatyny. Na poziomie globalnym zagęszczenie chromatyny jest również regulowane przez kowalencyjne modyfikacje histonów w nukleosomach.
Wykazano na przykład, że konstytutywna heterochromatyna (która nigdy nie jest wyrażana i jest gęsto upakowana) ma tendencję do przylegania do blaszki jądrowej, pozostawiając wolne pory jądra..
Ze swojej strony konstytutywna euchromatyna (która jest zawsze wyrażana, na przykład ta, która obejmuje geny utrzymania komórki i jest zlokalizowana w regionach luźnej chromatyny), robi to w dużych pętlach, które odsłaniają DNA do transkrypcji do mechanizmu transkrypcyjnego.
Inne regiony genomowego DNA oscylują między tymi dwoma stanami w zależności od czasu rozwoju organizmu, warunków wzrostu, tożsamości komórki itp..
Inne funkcje
Aby zrealizować swój plan rozwoju, ekspresji i utrzymania komórek, genomy organizmów eukariotycznych muszą precyzyjnie regulować, kiedy i jak muszą się manifestować ich potencjały genetyczne..
Począwszy od informacji przechowywanych w ich genach, są one zlokalizowane w jądrze w określonych regionach, które determinują ich stan transkrypcji..
Możemy zatem powiedzieć, że inną z fundamentalnych ról nukleosomów, poprzez zmiany w chromatynie, które pomaga to zdefiniować, jest organizacja lub architektura jądra, w którym się znajdują..
Ta architektura jest dziedziczona i chroniona filogenetycznie dzięki istnieniu tych modułowych elementów opakowania informacyjnego.
Bibliografia
- Alberts, B., Johnson, A. D., Lewis, J., Morgan, D., Raff, M., Roberts, K., Walter, P. (2014) Molecular Biology of the Cell (6th Wydanie). W. W. Norton & Company, Nowy Jork, NY, USA.
- Brooker, R. J. (2017). Genetyka: analiza i zasady. McGraw-Hill Higher Education, Nowy Jork, NY, USA.
- Cosgrove, M. S., Boeke, J. D., Wolberger, C. (2004). Regulowana ruchliwość nukleosomów i kod histonów. Nature Structural & Molecular Biology, 11: 1037–43.
- Goodenough, U. W. (1984) Genetics. W. B. Saunders Co. Ltd, Pkiladelphia, PA, USA.
- Griffiths, A. J. F., Wessler, R., Carroll, S. B., Doebley, J. (2015). Wprowadzenie do analizy genetycznej (11th red.). Nowy Jork: W. H. Freeman, Nowy Jork, NY, USA.


Jeszcze bez komentarzy