
Antykodon
Co to jest antykodon?
ZA antykodon to sekwencja trzech nukleotydów obecna w cząsteczce transferowego RNA (tRNA), której funkcją jest rozpoznanie kolejnej sekwencji trzech nukleotydów obecnej w cząsteczce informacyjnego RNA (mRNA).
To rozpoznawanie między kodonami i antykodonami jest antyrównoległe; to znaczy, że jeden znajduje się w kierunku 5 '-> 3', a drugi jest sprzężony w kierunku 3 '-> 5'. Rozpoznanie między trzema sekwencjami nukleotydów (trojaczkami) jest niezbędne dla procesu translacji; to znaczy w syntezie białek w rybosomie.
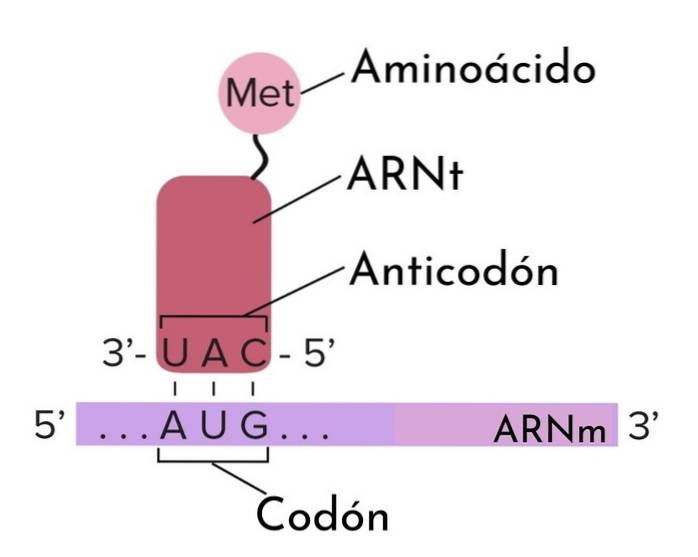
Zatem podczas translacji cząsteczki informacyjne RNA są „odczytywane” poprzez rozpoznawanie ich kodonów przez antykodony przenoszących RNA. Te cząsteczki są tak nazwane, ponieważ przenoszą określony aminokwas do cząsteczki białka, która powstaje na rybosomie..
Istnieje 20 aminokwasów, z których każdy jest kodowany przez określoną trójkę. Jednak niektóre aminokwasy są kodowane przez więcej niż jedną trójkę..
Dodatkowo, niektóre kodony są rozpoznawane przez antykodony w transferowych cząsteczkach RNA, które nie mają przyłączonych żadnych aminokwasów; są to tak zwane kodony stop.
Opis
Antykodon składa się z sekwencji trzech nukleotydów, które mogą zawierać dowolną z następujących zasad azotowych: adeninę (A), guaninę (G), uracyl (U) lub cytozynę (C) w połączeniu trzech nukleotydów, tak że działa jak kod.
Antykodony zawsze znajdują się w cząsteczkach transferowego RNA i zawsze znajdują się w odległości 3 '-> 5'. Struktura tych tRNA jest podobna do koniczyny, w taki sposób, że jest podzielona na cztery pętle (lub pętle); w jednej z pętli znajduje się antykodon.
Antykodony są niezbędne do rozpoznawania kodonów informacyjnych RNA, a tym samym do procesu syntezy białek we wszystkich żywych komórkach.
Funkcje antykodonu
Główną funkcją antykodonów jest specyficzne rozpoznawanie tripletów, które tworzą kodony w informacyjnych cząsteczkach RNA. Te kodony to instrukcje skopiowane z cząsteczki DNA w celu określenia kolejności aminokwasów w białku..
Ponieważ transkrypcja (synteza kopii informacyjnego RNA) zachodzi w kierunku 5 '-> 3', kodony informacyjne RNA mają tę orientację. Dlatego antykodony obecne w cząsteczkach przenoszącego RNA muszą mieć przeciwną orientację, 3 '-> 5'.
Ten związek jest wynikiem komplementarności. Na przykład, jeśli kodon to 5'-AGG-3 ', antykodonem jest 3'-UCC-5'. Ten rodzaj specyficznej interakcji między kodonami i antykodonami jest ważnym krokiem, który umożliwia sekwencji nukleotydów w informacyjnym RNA kodowanie sekwencji aminokwasów w białku..
Różnice między antykodonem a kodonem
- Antykodony to jednostki trinukleotydowe w tRNA, komplementarne do kodonów w mRNA. Umożliwiają one tRNA dostarczanie prawidłowych aminokwasów podczas produkcji białka. Zamiast tego kodony są jednostkami trinukleotydowymi w DNA lub mRNA, kodującymi określony aminokwas w syntezie białek..
- Antykodony są łącznikiem między sekwencją nukleotydów mRNA a sekwencją aminokwasów białka. Raczej kodony przenoszą informację genetyczną z jądra, w którym znajduje się DNA, do rybosomów, w których zachodzi synteza białek..
- Antykodon znajduje się w ramieniu antykodonu cząsteczki tRNA, w przeciwieństwie do kodonów, które znajdują się w cząsteczce DNA i mRNA..
- Antykodon jest komplementarny do odpowiedniego kodonu. Zamiast tego kodon w mRNA jest komplementarny do trypletu nukleotydów określonego genu w DNA..
- TRNA zawiera antykodon. Natomiast mRNA zawiera wiele kodonów.
Hipoteza huśtawki
Hipoteza swing zakłada, że połączenia między trzecim nukleotydem kodonu informacyjnego RNA a pierwszym nukleotydem antykodonu transferowego RNA są mniej specyficzne niż połączenia między dwoma pozostałymi nukleotydami trypletu..
Crick opisał to zjawisko jako „kołysanie” na trzeciej pozycji każdego kodonu. Coś dzieje się w tej pozycji, co pozwala na mniej rygorystyczne stawy niż zwykle. Jest również znany jako chybotanie lub chybotanie.
Ta hipoteza chybotania Cricka wyjaśnia, w jaki sposób antykodon danego tRNA może łączyć się z dwoma lub trzema różnymi kodonami mRNA..
Crick zasugerował, że skoro parowanie zasad (między podstawą 59 antykodonu w tRNA a zasadą 39 kodonu w mRNA) jest mniej ścisłe niż normalne, w tym miejscu dozwolone jest pewne „chybotanie” lub zmniejszone powinowactwo..
W rezultacie pojedynczy tRNA często rozpoznaje dwa lub trzy powiązane kodony, które określają dany aminokwas..
Zwykle wiązania wodorowe między zasadami antykodonów tRNA i kodonów mRNA podlegają ścisłym regułom parowania zasad tylko dla pierwszych dwóch zasad kodonu. Jednak efekt ten nie występuje we wszystkich trzecich pozycjach wszystkich kodonów mRNA..
RNA i aminokwasy
Na podstawie hipotezy chwiejności przewidziano istnienie co najmniej dwóch transferowych RNA dla każdego aminokwasu z kodonami wykazującymi całkowitą degenerację, co okazało się prawdą..
Ta hipoteza przewidywała również pojawienie się trzech transferowych RNA dla sześciu kodonów serynowych. Rzeczywiście, dla seryny scharakteryzowano trzy tRNA:
- TRNA dla seryny 1 (antykodon AGG) wiąże się z kodonami UCU i UCC.
- TRNA dla seryny 2 (antykodon AGU) wiąże się z kodonami UCA i UCG.
- TRNA dla seryny 3 (antykodon UCG) wiąże się z kodonami AGU i AGC.
Specyfika ta została zweryfikowana przez stymulowane wiązanie oczyszczonych trinukleotydów aminoacylo-tRNA z rybosomami in vitro..
Wreszcie, kilka transferowych RNA zawiera zasadową inozynę, która jest wytwarzana z hipoksantyny purynowej. Inozyna jest wytwarzana przez potranskrypcyjną modyfikację adenozyny.
Hipoteza chybotania Cricka przewidywała, że gdy inozyna jest obecna na końcu 5 'antykodonu (pozycja chybotania), sparuje się z uracylem, cytozyną lub adeniną w kodonie.
W rzeczywistości oczyszczony alanylo-tRNA zawierający inozynę (I) w pozycji 5 'antykodonu wiąże się z rybosomami aktywowanymi przez GCU, GCC lub GCA..
Ten sam wynik uzyskano z innymi tRNA oczyszczonymi inozyną w pozycji 5 'antykodonu. Dlatego hipoteza chwiejności Cricka bardzo dobrze wyjaśnia związki między tRNA a kodonami, biorąc pod uwagę kod genetyczny, który jest zdegenerowany, ale uporządkowany..
Bibliografia
- Brooker, R. (2012). Koncepcje genetyki (1st ed.). The McGraw-Hill Companies, Inc..
- Brązowy T. (2006). Genomy 3 (3r & D). Nauka o Garland.
- Griffiths, A., Wessler, S., Carroll, S. & Doebley, J. (2015). Wprowadzenie do analizy genetycznej (Wyd. 11). W.H. Obywatel
- Lewis, R. (2015). Genetyka człowieka: koncepcje i zastosowania(Wyd. 11). Edukacja McGraw-Hill.
- Snustad, D. & Simmons, M. (2011). Zasady genetyki(6th ed.). John Wiley and Sons.



Jeszcze bez komentarzy