
Historia DNA, funkcje, budowa, składniki
Plik DNA (kwas dezoksyrybonukleinowy) to biocząsteczka zawierająca wszystkie informacje niezbędne do wytworzenia organizmu i utrzymania jego funkcjonowania. Składa się z jednostek zwanych nukleotydami, składających się z grupy fosforanowej, pięciowęglowej cząsteczki cukru i zasady azotowej..
Istnieją cztery zasady azotowe: adenina (A), cytozyna (C), guanina (G) i tymina (T). Adenina zawsze łączy się w pary z tyminą, a guanina z cytozyną. Wiadomość zawarta w nici DNA jest przekształcana w informacyjny RNA, a ten bierze udział w syntezie białek.

DNA jest niezwykle stabilną cząsteczką, naładowaną ujemnie w fizjologicznym pH, która łączy się z białkami dodatnimi (histonami), aby skutecznie zagęszczać w jądrze komórek eukariotycznych. Długi łańcuch DNA wraz z różnymi związanymi z nim białkami tworzy chromosom.
Indeks artykułów
- 1 Historia
- 2 składniki
- 3 Struktura
- 3.1 Prawo Chargaffa
- 3.2 Model z podwójną helisą
- 4 Organizacja
- 4.1 Histony
- 4.2 Nukleosomy i włókno 30 nm
- 4.3 Chromosomy
- 4.4 Organizacja u prokariontów
- 4.5 Ilość DNA
- 5 Strukturalne formy DNA
- 5.1 DNA-A
- 5.2 Z-DNA
- 6 Funkcje
- 6.1 Replikacja, transkrypcja i translacja
- 6.2 Kod genetyczny
- 7 Właściwości chemiczne i fizyczne
- 8 Evolution
- 9 Sekwencjonowanie DNA
- 9.1 Metoda Sangera
- 10 Sekwencjonowanie nowej generacji
- 11 Odnośniki
Fabuła
W 1953 roku Amerykanin James Watson i Brytyjczyk Francis Crick zdołali wyjaśnić trójwymiarową strukturę DNA, dzięki pracom krystalograficznym wykonanym przez Rosalind Franklin i Maurice'a Wilkinsa. Swoje wnioski oparli również na pracach innych autorów.
Kiedy DNA jest wystawiane na promieniowanie rentgenowskie, tworzy się wzór dyfrakcyjny, który można wykorzystać do wywnioskowania struktury cząsteczki: helisa dwóch antyrównoległych łańcuchów, które obracają się w prawo, gdzie oba łańcuchy są połączone wiązaniami wodorowymi między zasadami. . Uzyskany wzór był następujący:

Strukturę można przyjąć zgodnie z prawami dyfrakcji Bragga: kiedy obiekt jest wstawiony w środku wiązki promieniowania rentgenowskiego, jest on odbijany, ponieważ elektrony obiektu oddziałują z wiązką..
25 kwietnia 1953 r. W prestiżowym magazynie opublikowano wyniki Watsona i Cricka Natura, w artykule zawierającym tylko dwie strony zatytułowanym „Struktura molekularna kwasów nukleinowych”, Co całkowicie zrewolucjonizowałoby dziedzinę biologii.
Dzięki temu odkryciu naukowcy otrzymali w 1962 roku Nagrodę Nobla w dziedzinie medycyny, z wyjątkiem Franklina, który zmarł przed porodem. Obecnie to odkrycie jest jednym z wielkich wykładników sukcesu naukowej metody zdobywania nowej wiedzy.
Komponenty
Cząsteczka DNA składa się z nukleotydów, jednostek składających się z pięciowęglowego cukru przyłączonego do grupy fosforanowej i zasady azotowej. Rodzaj cukru znajdującego się w DNA jest typem dezoksyrybozy i stąd jego nazwa, kwas dezoksyrybonukleinowy..
Aby utworzyć łańcuch, nukleotydy są kowalencyjnie połączone wiązaniem typu fosfodiestru poprzez grupę 3'-hydroksylową (-OH) z cukru i 5'-fosfafo następnego nukleotydu.
Nie należy mylić nukleotydów z nukleozydami. To ostatnie odnosi się do części nukleotydu utworzonej tylko przez pentozę (cukier) i zasadę azotową.
DNA składa się z czterech typów zasad azotowych: adeniny (A), cytozyny (C), guaniny (G) i tyminy (T).
Zasady azotowe dzielą się na dwie kategorie: puryny i pirymidyny. Pierwsza grupa składa się z pierścienia pięciu atomów połączonych z innym pierścieniem sześciu, podczas gdy pirymidyny składają się z pojedynczego pierścienia.
Spośród wymienionych zasad adenina i guanina pochodzą z puryn. Natomiast do grupy pirymidyn należą tymina, cytozyna i uracyl (obecne w cząsteczce RNA).
Struktura
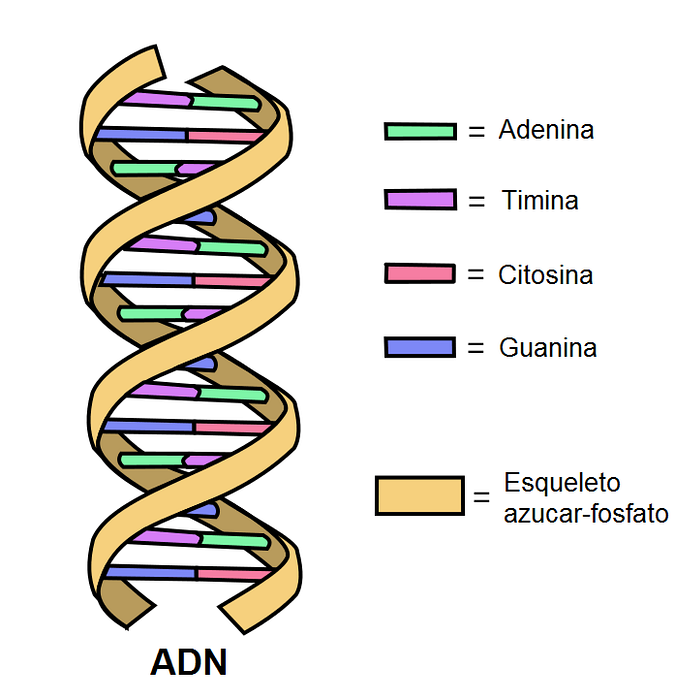
Cząsteczka DNA składa się z dwóch łańcuchów nukleotydów. Ten „łańcuch” jest znany jako nić DNA..
Dwie nici są połączone wiązaniami wodorowymi między uzupełniającymi się zasadami. Zasady azotowe są kowalencyjnie połączone ze szkieletem cukrów i fosforanów.
Każdy nukleotyd znajdujący się na jednej nici może być sprzężony z innym specyficznym nukleotydem na drugiej nici, tworząc dobrze znaną podwójną helisę. Aby utworzyć wydajną strukturę, A zawsze łączy się z T za pomocą dwóch wiązań wodorowych, a G z C za pomocą trzech mostków..
Prawo Chargaffa
Jeśli zbadamy proporcje zasad azotowych w DNA, okaże się, że ilość A jest identyczna z ilością T i taka sama z G i C. Ten wzór jest znany jako prawo Chargaffa.
To połączenie jest energetycznie korzystne, ponieważ pozwala zachować podobną szerokość w całej strukturze, zachowując podobną odległość wzdłuż cząsteczki szkieletu cukrowo-fosforanowego. Zauważ, że podstawa pierścienia łączy się z podstawą pierścienia.
Model z podwójną helisą
Sugeruje się, że podwójna helisa składa się z 10,4 nukleotydów na obrót, oddzielonych odległością od środka do środka wynoszącą 3,4 nanometra. Proces walcowania powoduje powstawanie rowków w konstrukcji, dzięki czemu można zaobserwować większy i mniejszy rowek.
Rowki powstają, ponieważ wiązania glikozydowe w parach zasad nie znajdują się naprzeciw siebie w odniesieniu do ich średnicy. Pirymidyna O-2 i puryna N-3 znajdują się w mniejszym rowku, podczas gdy główny rowek znajduje się w przeciwnym regionie..
Jeśli posłużymy się analogią drabiny, szczeble składają się z par uzupełniających się względem siebie podstaw, podczas gdy szkielet odpowiada dwóm poręczom..
Końce cząsteczki DNA nie są takie same, dlatego mówimy o „polarności”. Jeden z jego końców, 3 ', niesie grupę -OH, podczas gdy koniec 5' ma wolną grupę fosforanową.
Te dwie nici są położone przeciwrównolegle, co oznacza, że są położone przeciwnie do swoich biegunów, w następujący sposób:
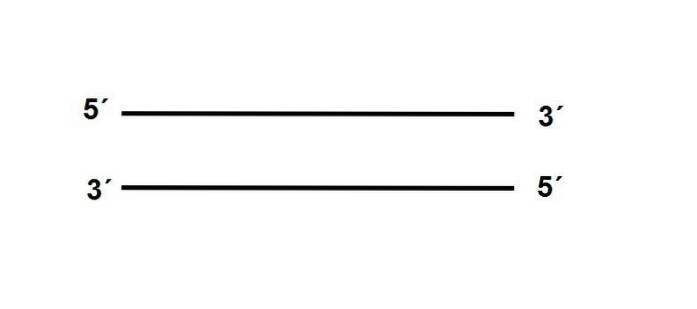
Ponadto sekwencja jednej z nici musi być komplementarna do jej partnera, jeśli jest to pozycja A, w nici antyrównoległej musi znajdować się T.
Organizacja
W każdej komórce ludzkiej znajduje się około dwóch metrów DNA, które należy skutecznie upakować.
Nić musi zostać zagęszczona tak, aby mogła znajdować się w mikroskopijnym jądrze o średnicy 6 μm, które zajmuje tylko 10% objętości komórki. Jest to możliwe dzięki następującym stopniom zagęszczenia:
Histony
U eukariotów występują białka zwane histonami, które mają zdolność wiązania się z cząsteczką DNA, będąc pierwszym stopniem zagęszczenia nici. Histony mają ładunki dodatnie, aby móc oddziaływać z ujemnymi ładunkami DNA, dostarczanymi przez fosforany.
Histony to białka tak ważne dla organizmów eukariotycznych, że praktycznie nie uległy zmianie w toku ewolucji - pamiętając, że niski wskaźnik mutacji wskazuje, że presja selekcyjna na tę cząsteczkę jest silna. Uszkodzenie histonu może prowadzić do wadliwego zagęszczenia DNA.
Histony można modyfikować biochemicznie, a proces ten modyfikuje stopień zagęszczenia materiału genetycznego.
Kiedy histony są „hipoacetylowane” chromatyna jest bardziej skondensowana, ponieważ formy acetylowane neutralizują dodatnie ładunki lizyn (dodatnio naładowanych aminokwasów) w białku..
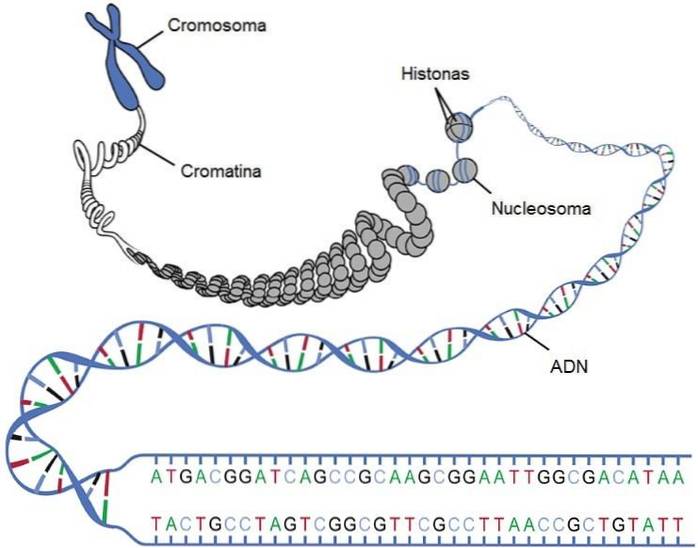
Nukleosomy i włókno 30 nm
Nić DNA zwija się w histony i tworzą struktury przypominające koraliki na naszyjniku z pereł, zwane nukleosomami. Sercem tej struktury są dwie kopie każdego typu histonu: H2A, H2B, H3 i H4. Połączenie różnych histonów nazywa się „oktamerem histonu”.
Oktamer jest otoczony przez około 146 par zasad, krążących mniej niż dwa razy. Ludzka diploidalna komórka zawiera około 6,4 x 109 nukleotydy zorganizowane w 30 milionów nukleosomów.
Organizacja w nukleosomach pozwala na zagęszczanie DNA do ponad jednej trzeciej jego pierwotnej długości.
W procesie ekstrakcji materiału genetycznego w warunkach fizjologicznych obserwuje się, że nukleosomy są ułożone w 30-nanometrowym włóknie.
Chromosomy
Chromosomy są funkcjonalną jednostką dziedziczności, której funkcją jest przenoszenie genów osobnika. Gen to odcinek DNA zawierający informacje potrzebne do syntezy białka (lub serii białek). Istnieją jednak geny kodujące elementy regulatorowe, takie jak RNA.
Wszystkie ludzkie komórki (z wyjątkiem gamet i czerwonych krwinek) mają dwie kopie każdego chromosomu, jedną odziedziczoną od ojca, a drugą od matki.
Chromosomy to struktury składające się z długiego liniowego fragmentu DNA związanego z wyżej wymienionymi kompleksami białkowymi. Zwykle u eukariontów cały materiał genetyczny zawarty w jądrze jest podzielony na serię chromosomów.
Organizacja u prokariotów
Prokariota to organizmy pozbawione jądra. U tych gatunków materiał genetyczny jest silnie zwinięty razem z alkalicznymi białkami o niskiej masie cząsteczkowej. W ten sposób DNA zostaje zagęszczone i zlokalizowane w centralnym regionie bakterii..
Niektórzy autorzy często nazywają tę strukturę „chromosomem bakteryjnym”, chociaż nie ma ona takich samych cech charakterystycznych jak chromosom eukariotyczny.
Ilość DNA
Nie wszystkie gatunki organizmów zawierają taką samą ilość DNA. W rzeczywistości wartość ta jest bardzo zmienna między gatunkami i nie ma związku między ilością DNA a złożonością organizmu. Ta sprzeczność jest znana jako „paradoks wartości C”.
Logicznym rozumowaniem byłoby przeczucie, że im bardziej złożony jest organizm, tym więcej ma DNA. Jednak nie jest to prawdą w naturze..
Na przykład genom lungfish Protopterus aethiopicus Ma 132 pg (DNA można określić ilościowo w pikogramach = pg), podczas gdy ludzki genom waży tylko 3,5 pg.
Należy pamiętać, że nie całe DNA organizmu koduje białka, duża ich część jest związana z elementami regulatorowymi i różnymi typami RNA..
Strukturalne formy DNA
Model Watsona i Cricka, wydedukowany z wzorców dyfrakcji rentgenowskiej, jest znany jako helisa B-DNA i jest „tradycyjnym” i najbardziej znanym modelem. Istnieją jednak dwie inne różne formy, zwane A-DNA i Z-DNA..
DNA-A
Wariant „A” skręca w prawo, podobnie jak B-DNA, ale jest krótszy i szerszy. Ta forma pojawia się, gdy wilgotność względna spada.
A-DNA obraca się co 11 par zasad, główny rowek jest węższy i głębszy niż B-DNA. W odniesieniu do mniejszego rowka jest to bardziej powierzchowne i szerokie.
Z-DNA
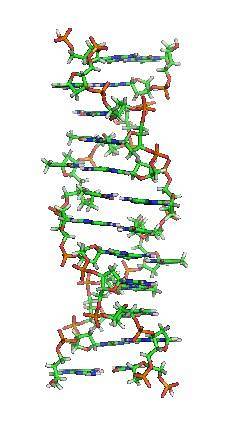
Trzeci wariant to Z-DNA. Jest to najwęższa forma, utworzona przez grupę heksanukleotydów zorganizowanych w dupleks antyrównoległych łańcuchów. Jedną z najbardziej wyróżniających się cech tego kształtu jest to, że skręca w lewo, podczas gdy pozostałe dwa sposoby robią to w prawo..
Z-DNA pojawia się, gdy występują na przemian krótkie sekwencje pirymidyn i puryn. Bruzda większa jest płaska, a mniejsza jest wąska i głębsza w porównaniu z B-DNA.
Chociaż w warunkach fizjologicznych cząsteczka DNA występuje głównie w formie B, istnienie dwóch opisanych wariantów uwidacznia elastyczność i dynamizm materiału genetycznego..
funkcje
Cząsteczka DNA zawiera wszystkie informacje i instrukcje niezbędne do budowy organizmu. Nazywa się pełny zestaw informacji genetycznej w organizmach genom.
Wiadomość jest kodowana przez „alfabet biologiczny”: cztery wspomniane wcześniej zasady: A, T, G i C.
Wiadomość może prowadzić do powstania różnych typów białek lub kodu dla jakiegoś elementu regulatorowego. Proces, za pomocą którego te bazy danych mogą dostarczyć wiadomość, został wyjaśniony poniżej:
Replikacja, transkrypcja i tłumaczenie
Wiadomość zaszyfrowana czterema literami A, T, G i C skutkuje fenotypem (nie wszystkie sekwencje DNA kodują białka). Aby to osiągnąć, DNA musi się replikować w każdym procesie podziału komórki..
Replikacja DNA jest półkonserwatywna: jedna nić służy jako szablon do tworzenia nowej cząsteczki potomnej. Replikację katalizują różne enzymy, w tym prymaza DNA, helikaza DNA, ligaza DNA i topoizomeraza..
Następnie wiadomość - napisana w języku sekwencji zasad - musi zostać przekazana do cząsteczki pośredniej: RNA (kwas rybonukleinowy). Ten proces nazywa się transkrypcją..
Aby doszło do transkrypcji, muszą uczestniczyć różne enzymy, w tym polimeraza RNA.
Enzym ten jest odpowiedzialny za kopiowanie wiadomości DNA i przekształcanie go w przekaźnikową cząsteczkę RNA. Innymi słowy, celem transkrypcji jest uzyskanie posłańca.
Wreszcie, dzięki rybosomom, następuje translacja wiadomości na informacyjne cząsteczki RNA.
Struktury te pobierają informacyjny RNA i razem z maszynerią translacyjną tworzą określone białko..
Kod genetyczny
Wiadomość czyta się w „trojaczkach” lub grupach po trzy litery, które określają aminokwas - element budulcowy białek. Odszyfrowanie przesłania trojaczków jest możliwe, ponieważ kod genetyczny został już całkowicie ujawniony.
Tłumaczenie zawsze zaczyna się od aminokwasu metioniny, który jest kodowany przez początkową trójkę: AUG. „U” oznacza podstawowy uracyl i jest charakterystyczny dla RNA i zastępuje tyminę.
Na przykład, jeśli informacyjny RNA ma następującą sekwencję: AUG CCU CUU UUU UUA, ulega translacji na następujące aminokwasy: metioninę, prolinę, leucynę, fenyloalaninę i fenyloalaninę. Zauważ, że dwie trojaczki - w tym przypadku UUU i UUA - mogą kodować ten sam aminokwas: fenyloalaninę.
Ze względu na tę właściwość mówi się, że kod genetyczny jest zdegenerowany, ponieważ aminokwas jest kodowany przez więcej niż jedną sekwencję tripletów, z wyjątkiem aminokwasu metioniny, który dyktuje rozpoczęcie translacji..
Proces jest zatrzymywany przez określone trojaczki typu stop lub stop: UAA, UAG i UGA. Znane są odpowiednio pod nazwami ochry, bursztynu i opalu. Kiedy rybosom je wykryje, nie mogą już dodawać kolejnych aminokwasów do łańcucha.
Właściwości chemiczne i fizyczne
Kwasy nukleinowe mają charakter kwaśny i są rozpuszczalne w wodzie (hydrofilowe). Może wystąpić tworzenie się wiązań wodorowych między grupami fosforanowymi i grupami hydroksylowymi pentoz z wodą. Przy fizjologicznym pH jest naładowany ujemnie.
Roztwory DNA są bardzo lepkie ze względu na odporność na odkształcenia podwójnej helisy, która jest bardzo sztywna. Lepkość spada, jeśli kwas nukleinowy jest jednoniciowy.
Są to wysoce stabilne cząsteczki. Logicznie rzecz biorąc, ta cecha musi być niezbędna w strukturach przenoszących informację genetyczną. W porównaniu z RNA, DNA jest dużo bardziej stabilne, ponieważ nie zawiera grupy hydroksylowej.
DNA może ulegać denaturacji termicznej, co oznacza, że nici rozdzielają się, gdy cząsteczka jest wystawiona na działanie wysokich temperatur.
Ilość ciepła, które należy zastosować, zależy od procentu G-C w cząsteczce, ponieważ te zasady są połączone trzema wiązaniami wodorowymi, co zwiększa odporność na separację..
Jeśli chodzi o absorpcję światła, mają one pik przy 260 nanometrach, który wzrasta, jeśli kwas nukleinowy jest jednoniciowy, ponieważ pierścienie nukleotydowe są odsłonięte i są odpowiedzialne za absorpcję..
Ewolucja
Według Lazcano et al. 1988 DNA wyłania się w fazie przejściowej z RNA, będąc jednym z najważniejszych wydarzeń w historii życia.
Autorzy proponują trzy etapy: pierwszy okres, w którym występowały cząsteczki podobne do kwasów nukleinowych, później genomy składały się z RNA, a na ostatnim etapie pojawiły się dwupasmowe genomy DNA..
Niektóre dowody potwierdzają teorię świata pierwotnego opartego na RNA. Po pierwsze, synteza białek może zachodzić przy braku DNA, ale nie wtedy, gdy brakuje RNA. Ponadto odkryto cząsteczki RNA o właściwościach katalitycznych..
Jeśli chodzi o syntezę dezoksyrybonukleotydów (obecnych w DNA), zawsze pochodzą one z redukcji rybonukleotydów (obecnych w RNA).
Ewolucyjna innowacja cząsteczki DNA musiała wymagać obecności enzymów syntetyzujących prekursory DNA i uczestniczących w odwrotnej transkrypcji RNA.
Badając obecne enzymy, można wywnioskować, że białka te ewoluowały kilkakrotnie i że przejście z RNA do DNA jest bardziej złożone niż wcześniej sądzono, w tym procesy przenoszenia i utraty genów oraz nieortologiczne zastąpienia..
sekwencjonowanie DNA
Sekwencjonowanie DNA polega na wyjaśnieniu sekwencji nici DNA pod względem czterech zasad, które ją tworzą.
Znajomość tej sekwencji ma ogromne znaczenie w naukach biologicznych. Może być używany do rozróżniania między dwoma gatunkami bardzo podobnymi morfologicznie, do wykrywania chorób, patologii lub pasożytów, a nawet ma zastosowanie w medycynie sądowej.
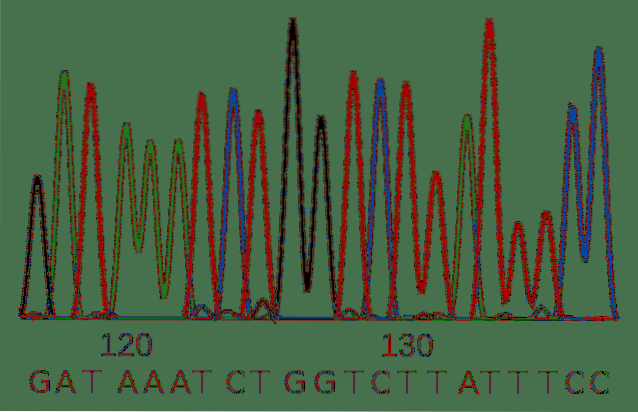
Sekwencjonowanie Sangera zostało opracowane w 1900 roku i jest tradycyjną techniką klarowania sekwencji. Mimo swojego wieku jest to sprawdzona metoda i szeroko stosowana przez badaczy.
Metoda Sangera
Metoda wykorzystuje polimerazę DNA, wysoce niezawodny enzym, który replikuje DNA w komórkach, syntetyzując nową nić DNA przy użyciu wcześniej istniejącej jako przewodnika. Enzym wymaga pierwszy lub starter do zainicjowania syntezy. Starter to mała cząsteczka DNA komplementarna do cząsteczki, która ma być sekwencjonowana.
W reakcji dodawane są nukleotydy, które mają zostać włączone do nowej nici DNA przez enzym.
Oprócz „tradycyjnych” nukleotydów, metoda obejmuje serię dideoksynukleotydów dla każdej z zasad. Różnią się od standardowych nukleotydów dwiema cechami: strukturalnie nie pozwalają polimerazie DNA na dodanie większej ilości nukleotydów do nici potomnej i mają inny marker fluorescencyjny dla każdej zasady.
Rezultatem jest różnorodność cząsteczek DNA o różnej długości, ponieważ dideoksynukleotydy zostały włączone losowo i zatrzymały proces replikacji na różnych etapach..
Tę różnorodność cząsteczek można rozdzielić ze względu na ich długość, a tożsamość nukleotydów odczytuje się za pomocą emisji światła ze znacznika fluorescencyjnego..
Sekwencjonowanie nowej generacji
Opracowane w ostatnich latach techniki sekwencjonowania pozwalają na masową analizę milionów próbek jednocześnie.
Do najwybitniejszych metod należą pirosekwencjonowanie, sekwencjonowanie przez syntezę, sekwencjonowanie przez ligację i sekwencjonowanie nowej generacji przez Ion Torrent..
Bibliografia
- Alberts, B., Johnson, A., Lewis, J. i in. (2002). Biologia molekularna komórki. Wydanie 4. Nowy Jork: Garland Science. Struktura i funkcja DNA. Dostępne pod adresem: ncbi.nlm.nih.gov/
- Alberts, B., Johnson, A., Lewis, J. i in. (2002). Biologia molekularna komórki. Wydanie 4. Nowy Jork: Garland Science. Chromosomalne DNA i jego opakowanie we włóknie chromatyny. Dostępne pod adresem: ncbi.nlm.nih.gov
- Berg, J.M., Tymoczko, J.L., Stryer, L. (2002). Biochemia. Wydanie 5. Nowy Jork: W H Freeman. Sekcja 27.1, DNA może przybierać różne formy strukturalne. Dostępne pod adresem: ncbi.nlm.nih.gov
- Fierro, A. (2001). Krótka historia odkrycia struktury DNA. Rev Méd Clínica Las Condes, dwadzieścia, 71-75.
- Forterre, P., Filée, J. & Myllykallio, H. (2000–2013) Origin and Evolution of DNA and DNA Replication Machineries. Na: Baza danych Madame Curie Bioscience [Internet]. Austin (Teksas): Landes Bioscience. Dostępne pod adresem: ncbi.nlm.nih.gov
- Lazcano, A., Guerrero, R., Margulis, L. i Oro, J. (1988). Ewolucyjne przejście od RNA do DNA we wczesnych komórkach. Dziennik ewolucji molekularnej, 27(4), 283-290.
- Lodish, H., Berk, A., Zipursky, S.L., et al. (2000). Molecular Cell Biology. Wydanie 4. Nowy Jork: W. H. Freeman. Sekcja 9.5, Organizowanie DNA komórkowego w chromosomy. Dostępne pod adresem: ncbi.nlm.nih.gov/books
- Voet, D., Voet, J. G. i Pratt, C. W. (1999). Podstawy biochemii. Nowy York: John Willey and Sons.


Jeszcze bez komentarzy